- Elena Kardash Ph. D.eekardash at gmail dot comMax Planck Institute for Molecular Biomedicine, Muenster, Germany
- Galet Colette Ph. D.cogalet at gmail dot comUniversité de Californie à Los Angeles, États-Unis
Le poisson zébre (Danio Rerio) est un poisson d’eau douce qui vit dans les rivières de l’Inde, du Pakistan et autres pays d’Asie. Au cours des deux dernières décennies, le poisson zébre est devenu l’un des organismes modèles in vivo pour l'étude de divers processus tels que l'embryogenèse, la migration cellulaire, la formation des organes, le comportement et le sommeil. Récemment, le poisson zèbre a suscité l'intérêt dans d'autres branches de la recherche biomédicale en raison de son potentiel émergent pour la découverte de médicaments dans différents modèles de maladies. Cette revue présente une sélection des méthodes les plus couramment utilisées dans la recherche sur le poisson zèbre. Alors que l'élevage et la manipulation du poisson zèbre sont détaillés dans "Zebrafish Book" [The zebrafish Book http://zfin.org/zf_info/zfbook/cont.html], cet article présente les outils importants et les méthodologies disponibles dans la recherche sur le poisson zèbre comme la micro-injection, le morpholino knockdown et, la transplantation, discute leurs applications.
Le poisson zèbre est devenu un modèle de choix dans la recherche biomédicale portant sur un large éventail de sujets allant de la biologie du développement et de la morphogenèse aux neurosciences, la régénération et le vieillissement au cours des dernières décennies. La facilité d'entretien, le grand nombre de descendants, une option permettant la sélection génétique, et la clarté optique des embryons en développement constituent quelques facteurs qui rendent le poisson zèbre un modèle attrayant. Alors que le principal avantage du poisson zèbre est la possibilité de procéder à de l'imagerie in vivo haute résolution, la communauté a pris du retard dans le développement d’animaux transgéniques et pour le « knock-out » efficace de gènes par rapport à d’autres modèles comme la drosophile (Drosophila melanogaster) et la souris en raison d'un manque d'outils génétiques appropriés. Cependant, au cours des dernières années, ces écarts se sont réduits en raison de la progression rapide de la technologie. De nouveaux outils tels que les nucléases à doigts de zinc qui permettent un « knock-out » efficace de gènes ou les technologies MAZe et brainbow pour la création d’animaux transgéniques, permettent d'évaluer la complexité des processus de développement et d’acquérir une meilleure compréhension des mécanismes des maladies chez un vertébré.
De plus, les génomes de l'homme et du poisson zèbre partagent environ 70 % d'homologie, suggérant la conservation d’un grand nombre de gènes et de voies génétiques. En raison de la disponibilité des plates-formes technologiques, le poisson zèbre a été utilisé dans la sélection de médicaments à débit moyen dans le contexte de l'organisme complet (par rapport aux systèmes acellulaires ou de cellule unique). Cette revue se concentre sur les méthodes pertinentes, à commencer par les méthodes d’injection, d’immunohistologie et le marquage in situ, puis discute le « knock down » de l’expression des gènes, la mutagénèse ciblée, les outils transgéniques, et les expériences de mosaïque.
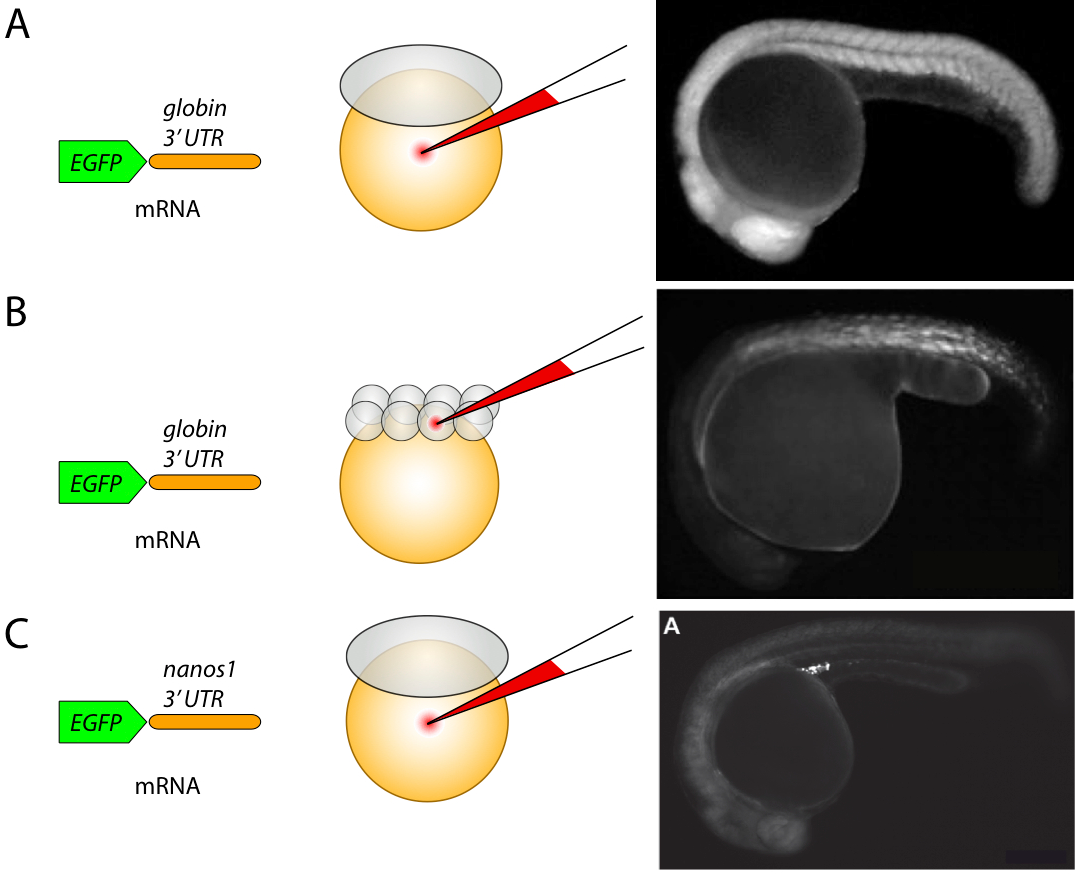
L'injection est la technique la plus fréquemment utilisée lorsque l'on travaille avec le poisson zèbre. Pendant l'injection, une certaine substance comme l'acide nucléique, une protéine ou une drogue est introduite dans l'embryon, le plus souvent dans les premiers stades de développement (Figure 1). Les injections sont effectuées avec des aiguilles de verre remplies avec le matériel à délivrer. Des micromanipulateurs contrôlent normalement les aiguilles et peuvent changer la position d'une aiguille par rapport à l'embryon avec facilité. La pression d'air fournie par un dispositif externe est utilisée pour libérer le contenu de l’aiguille dans l'embryon. L'injection permet une livraison rapide du matériel dans les embryons. Un chercheur expérimenté peut injecter quelques centaines d'embryons en une heure. Un protocole d'injection détaillée est disponible dans la référence [1] et ses films peuvent être consultés sur [Microinjection of Zebrafish Embryos to Analyze Gene Function http://www.jove.com/details.php?id=1115].
Les injections sont le plus souvent utilisées pour la surexpression transitoire de protéines dans les embryons. La surexpression transitoire pendant le développement précoce du poisson zèbre (jusqu'à 3 jours) est réalisée par l'introduction de l'ARNm codant pour la protéine d'intérêt dans les embryons au cours des deux premières heures de développement. Pour stabiliser la transcription de l'ARNm injecté, les régions d'ARNm régulatrices, telles que les régions non traduites (UTR) en 3’ sont utilisées (Figure 1). Il s'agit notamment des séquences poly A tels que celles présentes dans la beta- globine de Xenopus laevis, qui stabilisent la transcription de l'ARNm dans chaque cellule de l'embryon contenant cet ARNm (figure 1A et B). Pour certains types de cellules, comme les cellules germinales primordiales, des régions UTRs spécifiques telles que celles trouvées dans le 3’ -UTR du gène Nanos1 peuvent être utilisées pour stabiliser l'ARNm et pour promouvoir la traduction efficace des protéines, spécifiquement dans ces cellules tout en le dégradant dans les cellules somatiques [2] (figure 1C). Un autre exemple montrant le rôle des régions 3’ -UTR dans la localisation de l'ARNm est l'ARNm de strabisme, qui est confiné dans les blastomères dorsaux dès le stade de quatre cellules [3].
Pour diriger l'expression de la protéine dans chaque cellule de l'embryon en développement, l'ARNm correspondant à une protéine spécifique est injecté dans le sac vitellin de l'embryon au stade une cellule (figure 1A). Pour générer des embryons mosaïques, dans lesquels la protéine est exprimée dans une sous-population de cellules, l'ARNm est injecté dans un seul blastomère de l'embryon au stade 8 cellules ou dans les étapes ultérieures (figure 1B). Une autre stratégie utilisée pour obtenir l'expression mosaïque d’une protéine est d’injecter les plasmides d'ADN dans un seul blastomère de l'embryon au stade une cellule ou plus tard (directement dans une cellule spécifique), mais cette approche est moins efficace et prend plus de temps pour la synthèse des protéines par rapport à l'injection de l'ARNm.
Bien que l'expression transitoire de protéines par injection d'ARNm soit un moyen rapide et facile d'étudier la localisation des protéines et la fonction chez le poisson zèbre dans les premiers stades de développement, cette approche ne convient pas lorsque l’on s’intéresse aux derniers stades de développement ou lorsqu'une protéine doit être exprimée dans un tissu particulier ou un type de cellule.
La technique d'hybridation in situ est une des plus anciennes méthodes utilisées dans la recherche sur le poisson zèbre. Elle permet l’analyse de profils d'expression des gènes dans les embryons intacts ou sectionnés [4]. Au cours de la procédure dans l'hybridation in situ, une sonde d'ARNm antisens est conçue pour reconnaître et lier le transcrit endogène, qui est ensuite détecté par une technique basée sur la couleur ou la fluorescence. L'approche classique utilise une procédure de marquage basée sur la couleur et le signal est visualisé à l'aide d'un microscope optique (figure 2A). Récemment, les techniques de détection fluorescente ont été optimisées pour l'utilisation chez le poisson zèbre, permettant la détection simultanée de trois sondes avec l'approche normale, ou jusqu'à cinq sondes en utilisant l'amplification HCR (figure 2B et C) [5] [6]. L’hybridation in situ basée sur la fluorescence est la méthode de détection du signal préférée en raison de sa sensibilité accrue et de la détection simultanée de plusieurs sondes, tandis que la détection basée sur la couleur se limite à deux sondes. Cependant, la détection basée sur la couleur classique ne nécessite pas de microscope confocal et peut être utilisée lorsque le microscope confocal n'est pas disponible.

L’immunomarquage est un procédé efficace pour détecter la présence et la localisation d'une protéine endogène. Le principal obstacle à l'exécution d'un immunomarquage réussi est l’exposition de l'antigène (« antigen retrieval »). Une autre difficulté provient d'une quantité relativement faible d'anticorps de haute qualité disponibles dans le commerce développés pour l'utilisation chez le poisson zèbre. Une étude récente a proposé une méthode améliorée pour le traitement des embryons pour l’exposition d'antigène efficace (figure 2D) [7].
Un certain nombre d'outils pour la transgénèse ont été adaptés à partir d'autres organismes et optimisés pour l'utilisation chez le poisson zèbre permettant l'expression stable de protéines et le ciblage conditionnel de protéines pour l'expression dans les tissus définis.
Le système le plus populaire pour la production d'animaux transgéniques chez le poisson zèbre est le système transposon Tol2, provenant du poisson medaka [8] [9]. Le produit de construction pour l'introduction de l'ADN dans le génome du poisson zèbre se compose généralement de deux séquences régulatrices cis minimale de l'élément Tol2 positionnées aux extrémités 5’ et 3’ d'un promoteur minimal suivi par une protéine fluorescente (figure 3). Le vecteur contenant Tol2 est apte à recevoir des inserts d'ADN allant jusqu’à 11 kb [8]. Une telle construction d'ADN est injectée conjointement avec l'ARNm codant pour un gène transposase directement dans la cellule de l'embryon au stade une cellule (figure 3). Ces embryons sont élevés et les poissons adultes sont examinés pour identifier les fondateurs, dans lequel les insertions ont été créées dans la lignée germinale qui peut être transmise à la génération suivante (représentée comme F1 dans la figure 3).

Une autre stratégie pour l'expression de protéines régulée qui a récemment été adaptée pour l'utilisation dans le poisson zèbre, est la recombinaison médiée par les sites Cre spécifiques [ref ref 10]. La recombinaison médiée par Cre permet l'induction de l'expression de la protéine d'une manière contrôlée temporellement (figure 4A). L'expression de la Cre-recombinase peut encore être régulée par le promoteur « Heat shock » permettant une haute précision spatiale et temporelle de l’expression de la protéine d’intérêt. Cette stratégie a été utilisée dans le système transgénique MAZe pour une expression de protéines spatio-temporelle dans les embryons mosaïques [11] (figure 4B). Le système MAZe combine l'utilisation de la Cre recombinase exprimée sous le promoteur « Heat shock » et le système GAL4/UAS adapté pour l'utilisation chez le poisson zèbre. Suite à l'induction de choc thermique, la Cre-recombinase est exprimée, induisant l'excision de la cassette fermée par les éléments lox P, ce qui permet la synthèse de la protéine Gal4VP16 qui à son tour se lie au promoteur UAS dirigeant l'expression de la protéine spécifique. Cette méthode est particulièrement utile lors de l'induction de l'expression de dominants négatifs dans une population choisie de cellules, à un temps défini.

Morpholinos. L'approche standard pour bloquer spécifiquement la fonction d'un gène dans un embryon de poisson zèbre dans les premiers stades de développement utilise des oligonucléotides antisens modifiés appelés morpholinos [12] [13]. Les morpholinos sont des molécules synthétiques, d’environ 25 nucléotides, et sont disponibles à Gene Tools [ref Gene Tools LLC http://www.gene-tools.com/].Deux types de morpholinos sont couramment utilisés pour interférer avec l'expression des protéines: les morpholinos ATG et les morpholinos d’épissage. Les morpholinos ATG agissent en bloquant l'initiation de la traduction des protéines dans les ribosomes, rendant ainsi les embryons dépourvus d'une protéine particulière [14]. La figure 5A montre un exemple d'un embryon de trois jours manquant ses nageoires pectorales à la suite d'un traitement avec le morpholino contre le gène fgf24 [15]. Les morpholinos d’épissage se lient et interférent avec la procédure d'épissage de l'ARN, résultant en une protéine tronquée. Par conséquent, les morpholinos d’épissage pourraient être aussi utilisés pour étudier la fonction d'un domaine protéique particulier.

Des morpholinos protecteurs de cibles ont été récemment développés pour interférer avec le fonctionnement des micro ARNs endogènes (miARN) en protégeant spécifiquement leurs cibles. Ces morpholinos sont complémentaires aux séquences de liaison des miARNs présentes dans les ARNm cibles et agissent en empêchant la liaison du miRNA à sa cible, stabilisant ainsi la transcription de l'ARNm [16]. De même, des morpholinos peuvent être utilisés pour inhiber la fonction des miARNs directement [17]. Les considérations générales dans le traitement des morpholinos et l'interprétation des résultats expérimentaux sont décrits en détail dans [14] [18].
Les morpholinos représentent une stratégie très efficace pour interférer avec un gène ou la fonction de miARN avec une grande précision, mais il y a plusieurs problèmes concernant morpholinos qui doivent être pris en considération.
- Les morpholinos sont normalement injectés dans le sac vitellin et pourraient donc également affecter toutes les cellules de l'embryon en développement et, ainsi, compromettre le bon développement dans les cas où la fonction de la protéine perturbée est nécessaire ubiquitairement. Ce problème peut cependant être évité par l'injection du morpholino dans un seul blastomère (figure 1B) dans le but de cibler plus précisément les cellules d'intérêt.
- L'effet de morpholinos ne dure que les quelques premiers jours de développement (jusqu'à 5 jours [14]), les rendant impropres à l’étude de la fonction d'un gène à des stades ultérieurs de développement.
- Les morpholinos ne sont pas efficaces lorsque la protéine maternelle apparentée est présente.
- Bien que les morpholinos éliminent la fonction des protéines, l'ARNm correspondant reste présent dans l'embryon. Dans les cas où l'ARNm joue un autre rôle en plus de servir de modèle pour la traduction des protéines, il pourrait potentiellement conduire à des effets non spécifiques, surtout si les taux d'ARNm sont importants.
Nucléases à doigts de zinc. Alors que, jusqu'à récemment, il n'existait pas de méthode fiable pour induire la mutagénèse ciblée chez le poisson zèbre, deux articles publiés en 2008 ont adapté l'utilisation des nucléases à doigts de zinc (ZFN) pour créer les cassures double brin ciblées dans le génome du poisson zèbre. Dans deux études indépendantes, les auteurs ont démontré l'utilisation des ZFNs pour générer des cassures, qui sont ensuite réparées par liaison d’extrémités non-homologues, résultant dans de petites insertions et des suppressions. ZFN représente une protéine de fusion chimérique comprenant une protéine à doigt de zinc (ZFP) et le domaine de clivage de l'endonucléase FokI [19]. La spécificité de liaison à l'ADN est définie par la ZFP, qui peut être conçue pour reconnaître une variété de séquences d'ADN cibles [19] (Figure 5B et [20], [21]). La partie importante dans la conception ZFN est l'optimisation de la protéine ZFP pour la reconnaissance de la cible. Des protocoles détaillés et des informations complémentaires sont disponibles sur le site ZFIN [Zinc Finger Consortium http://zincfingers.org/default2.htm] et des conseils sur la façon d’utiliser la plate-forme OPEN pour l'ingénierie d'un array de protéine ZFP sont donnés dans [22]. Des informations supplémentaires sur la façon de localiser des sites cibles ZFN peuvent être obtenues dans [23].

La transplantation est l'une des techniques les plus anciennes utilisées en biologie du développement. Au cours de la transplantation, un groupe de cellules est supprimée d’un embryon, appelé l'embryon donneur, et introduit dans un autre embryon, qui est appelé l'embryon accepteur (figure 6A). La technique de transplantation est essentielle pour répondre à certaines questions importantes en biologie du développement, comme savoir si une fonction génique est autonome ou non. Dans les expériences qui étudient la fonction des gènes, les cellules mutantes sont transplantées dans un embryon de type sauvage et, inversement, les cellules de type sauvage sont transplantées dans un embryon mutant et les phénotypes des cellules transplantées et ceux des embryons hôtes sont comparés. La transplantation peut également être utilisée lorsque l'injection de morpholinos est mortelle. Dans ce cas, un groupe de cellules de l'embryon injecté avec un morpholino est transplanté dans un embryon de type sauvage dans les premiers stades du développement embryonnaire, permettant ainsi d’étudier l'effet de l'inactivation de la protéine sur les cellules dans un environnement de type sauvage. Les transplantations sont également utilisées pour étudier l'effet d'un facteur diffusible, par exemple un facteur de croissance ou une chimiokine sur des cellules voisines. Un exemple de la façon dont les transplantations sont utilisées pour étudier les mécanismes qui sous-tendent les interactions neurone-glie est présentée dans [Tissue Targeted Embryonic Chimeras: Zebrafish Gastrula Cell Transplantation http://www.jove.com/details.php?id=1422] [24] (figure 6B).
- Rosen J, Sweeney M, Mably J. Microinjection of zebrafish embryos to analyze gene function. J Vis Exp. 2009;: PMID 19274045 CrossRef
- Köprunner M, Thisse C, Thisse B, Raz E. A zebrafish nanos-related gene is essential for the development of primordial germ cells. Genes Dev. 2001;15:2877-85 PMID 11691838
- Gore A, Maegawa S, Cheong A, Gilligan P, Weinberg E, Sampath K. The zebrafish dorsal axis is apparent at the four-cell stage. Nature. 2005;438:1030-5 PMID 16355228
- Jowett T, Lettice L. Whole-mount in situ hybridizations on zebrafish embryos using a mixture of digoxigenin- and fluorescein-labelled probes. Trends Genet. 1994;10:73-4 PMID 8178366
- Choi H, Chang J, Trinh L, Padilla J, Fraser S, Pierce N. Programmable in situ amplification for multiplexed imaging of mRNA expression. Nat Biotechnol. 2010;28:1208-12 PMID 21037591 CrossRef
- Lauter G, Söll I, Hauptmann G. Multicolor fluorescent in situ hybridization to define abutting and overlapping gene expression in the embryonic zebrafish brain. Neural Dev. 2011;6:10 PMID 21466670 CrossRef
- Inoue D, Wittbrodt J. One for all--a highly efficient and versatile method for fluorescent immunostaining in fish embryos. PLoS ONE. 2011;6:e19713 PMID 21603650 CrossRef
- Kawakami K. Tol2: a versatile gene transfer vector in vertebrates. Genome Biol. 2007;8:S7 PMID 18047699
- Urasaki A, Asakawa K, Kawakami K. Efficient transposition of the Tol2 transposable element from a single-copy donor in zebrafish. Proc Natl Acad Sci U S A. 2008;105:19827-32 PMID 19060204 CrossRef
- Hans S, Kaslin J, Freudenreich D, Brand M. Temporally-controlled site-specific recombination in zebrafish. PLoS ONE. 2009;4:e4640 PMID 19247481 CrossRef
- Collins R, Linker C, Lewis J. MAZe: a tool for mosaic analysis of gene function in zebrafish. Nat Methods. 2010;7:219-23 PMID 20139970 CrossRef
- Summerton J. Morpholino antisense oligomers: the case for an RNase H-independent structural type. Biochim Biophys Acta. 1999;1489:141-58 PMID 10807004
- Nasevicius A, Ekker S. Effective targeted gene 'knockdown' in zebrafish. Nat Genet. 2000;26:216-20 PMID 11017081
- Bill B, Petzold A, Clark K, Schimmenti L, Ekker S. A primer for morpholino use in zebrafish. Zebrafish. 2009;6:69-77 PMID 19374550 CrossRef
- Manfroid I, Delporte F, Baudhuin A, Motte P, Neumann C, Voz M, et al. Reciprocal endoderm-mesoderm interactions mediated by fgf24 and fgf10 govern pancreas development. Development. 2007;134:4011-21 PMID 17942484
- Choi W, Giraldez A, Schier A. Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430. Science. 2007;318:271-4 PMID 17761850
- Flynt A, Li N, Thatcher E, Solnica-Krezel L, Patton J. Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate. Nat Genet. 2007;39:259-63 PMID 17220889
- Eisen J, Smith J. Controlling morpholino experiments: don't stop making antisense. Development. 2008;135:1735-43 PMID 18403413 CrossRef
- Porteus M, Carroll D. Gene targeting using zinc finger nucleases. Nat Biotechnol. 2005;23:967-73 PMID 16082368
- Doyon Y, McCammon J, Miller J, Faraji F, Ngo C, Katibah G, et al. Heritable targeted gene disruption in zebrafish using designed zinc-finger nucleases. Nat Biotechnol. 2008;26:702-8 PMID 18500334 CrossRef
- Meng X, Noyes M, Zhu L, Lawson N, Wolfe S. Targeted gene inactivation in zebrafish using engineered zinc-finger nucleases. Nat Biotechnol. 2008;26:695-701 PMID 18500337 CrossRef
- Foley J, Maeder M, Pearlberg J, Joung J, Peterson R, Yeh J. Targeted mutagenesis in zebrafish using customized zinc-finger nucleases. Nat Protoc. 2009;4:1855-67 PMID 20010934 CrossRef
- Reyon D, Kirkpatrick J, Sander J, Zhang F, Voytas D, Joung J, et al. ZFNGenome: a comprehensive resource for locating zinc finger nuclease target sites in model organisms. BMC Genomics. 2011;12:83 PMID 21276248 CrossRef
- Deschene E, BARRESI M. Tissue targeted embryonic chimeras: zebrafish gastrula cell transplantation. J Vis Exp. 2009;: PMID 19749688 CrossRef
- Ellingsen S, Laplante M, König M, Kikuta H, Furmanek T, Hoivik E, et al. Large-scale enhancer detection in the zebrafish genome. Development. 2005;132:3799-811 PMID 16049110
- Tissue Targeted Embryonic Chimeras: Zebrafish Gastrula Cell Transplantation. Available from: www.jove.com/details.php?id=1422
- ZFIN ID: ZDB-IMAGE-070503-669. Available from: zfin.org/cgi-bin/webdriver?MIval=aa-imageview.apg&image_table=image&OID=ZDB-IMAGE-070503-669
